Nat Commun:北大高歌团队开发新型深度学习模型DECIPHER,破解细胞分子与空间背景“纠缠”难题
时间:2025-09-01 12:13:41 热度:37.1℃ 作者:网络
在多细胞生物中,细胞通过相互作用形成三维组织结构;因此,单个细胞的生理功能及潜在的病理改变不仅由其内在分子特性决定,还与其空间环境密切相关。目前,空间组学技术的发展使人们能够系统性地表征这两个维度的信息,但如何在计算机中准确呈现这些数据仍是一项重大挑战。
为应对这一挑战,当前主流方法多采用"整体嵌入"技术路径,即将细胞的如转录组信息与空间位置信息共同编码至统一低维空间中,形成综合性表征,以便于空间区域聚类、批次效应校正、多切片对齐等后续分析。这类方法虽简化了模型设计与实现过程,也使分子特征与空间信息出现固有“纠缠” ,难以解析其相互作用;无法兼容细胞间通讯分析中常用的非嵌入分析方法;以及存在计算可扩展性严重不足等情况。
近日,北京大学生物医学前沿创新中心(BIOPIC)高歌团队开发了一种新型深度学习模型DECIPHER,专门用于大规模、异质性空间组学数据的分析。该模型通过跨尺度对比学习策略,将细胞内分子特征与细胞外空间背景信息解耦,分别生成独立的嵌入表示。评估显示,DECIPHER在多个真实数据集和模拟数据上均优于当前主流方法,可解析多尺度下的细胞-环境相互作用;并具备高度可扩展性,能够处理包含数百万细胞的空间图谱。此外,该模型还能够识别与细胞空间定位相关的关键基因及配体-受体对,为空间组学数据分析提供了新工具。

与传统空间组学方法的整体嵌入表征不同,DECIPHER采用双编码器架构,以保留基因表达和空间环境中的共享信息和特定信息。其中,“组学编码器”由多层感知机(MLP)构成,可从基因表达谱提取细胞内在分子特征;“空间编码器”则基于Transformer架构构建,处理细胞之间的空间关系,提取空间背景嵌入。
两个组件通过专门设计的跨尺度对比学习机制实现同步优化,该机制不仅通过视图增强技术分别强化了分子及空间特征的表示学习,还兼具消除批次效应的能力。这种设计使得模型最终能产生解耦的双重嵌入表示——分子特征嵌入适用于传统组学分析任务,空间位置嵌入用于空间域识别等空间任务,从而实现了真正意义上的特征解耦与任务特异性优化。
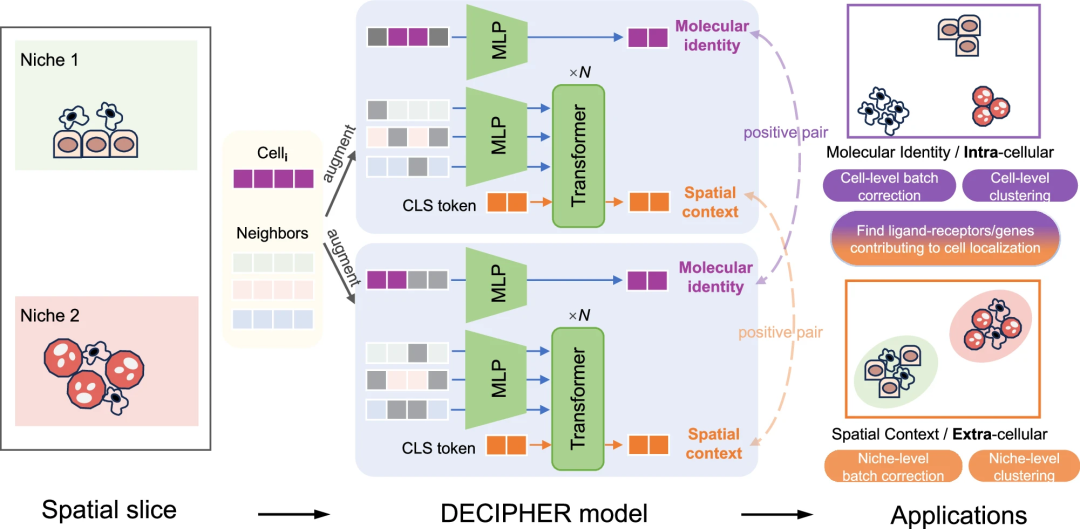
图1. DECIPHER概述
研究团队在Xenium人乳腺癌数据集、MERFISH小鼠脑数据集和使用两个10x PBMC数据集生成的模拟合成空间数据集中,将DECIPHER与Banksy、STAGATE、scVI、STADIA等多种基于不同策略的主流方法进行系统比较;以归一化互信息(NMI)、调整兰德指数(ARI)作为评估指标。
基准测试结果显示,无论是模拟数据集还是真实数据集中,DECIPHER的空间嵌入始终显著优于其他方法,与真实空间区域标注的一致性较高。在组学任务上,DECIPHER的解耦设计也有效避免了传统整体嵌入模型性能退化的问题。
更重要的是,DECIPHER模型还成功解决了制约领域发展的可扩展性瓶颈——多项主流GNN方法(GraphST、BASS、STAGATE等)因内存溢出或计算超时而无法处理百万级细胞数据集,而DECIPHER凭借Transformer架构实现了高效计算。在批次效应校正方面,DECIPHER同样展现出一流的性能,显著优于专用批次校正方法。这些结果充分证明,DECIPHER通过创新的解耦表征学习框架,在保持计算效率的同时,实现了对空间组学数据多维度信息的精准解析与整合。
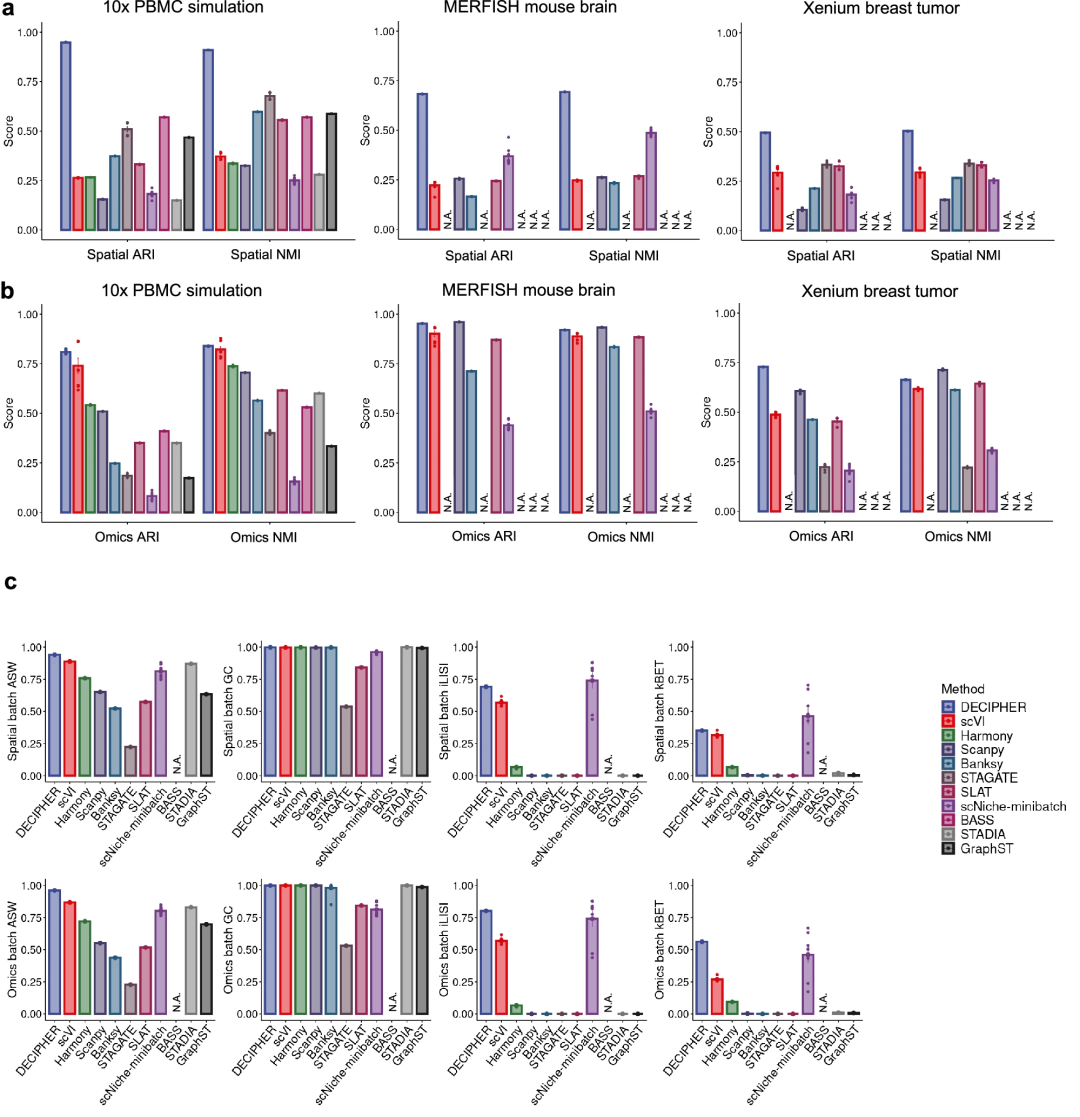
图2.DECIFYR和最先进方法的基准测试
细胞与环境的相互作用通常通过配体-受体(LR)对调控细胞定位,并进一步推动生物体发育与内环境稳态维持。研究团队分析了DECIPHER模型能否利用解耦后的嵌入,识别与细胞空间微环境密切相关的LR对或基因。
在淋巴结Xenium 5k数据集中,研究团队探究了影响B细胞在生发中心(GC)定位的关键细胞间通信(CCC)分子。结果显示,DECIPHER精准识别出CXCL12_CXCR4和CXCL13_CXCR5这两个已知LR对,其对B细胞成熟和定位至关重要;而NicheNet、CellChatV2 及传统DEG分析未能准确识别。此外,在一个已广泛研究的人类皮肤数据集中,DECIPHER也准确识别了重要的LR对GAS6-TYRO3和PROS1-TYRO3,这与NicheNet的结果一致。
研究团队还评估了DECIPHER在基因检测受限的场景中的性能。在仅覆盖313个基因的乳腺癌Xenium数据集中,DECIPHER仍能通过高保真嵌入成功识别出PTGDS(已证实为浸润性淋巴细胞关键标志物),以及CXCL12、CD86等多个与淋巴细胞活化募集相关的基因。这表明DECIPHER在识别细胞定位相关分子时,不仅准确性高,还能克服基因表达水平低、检测基因数量有限等场景的限制,显著优于传统方法。
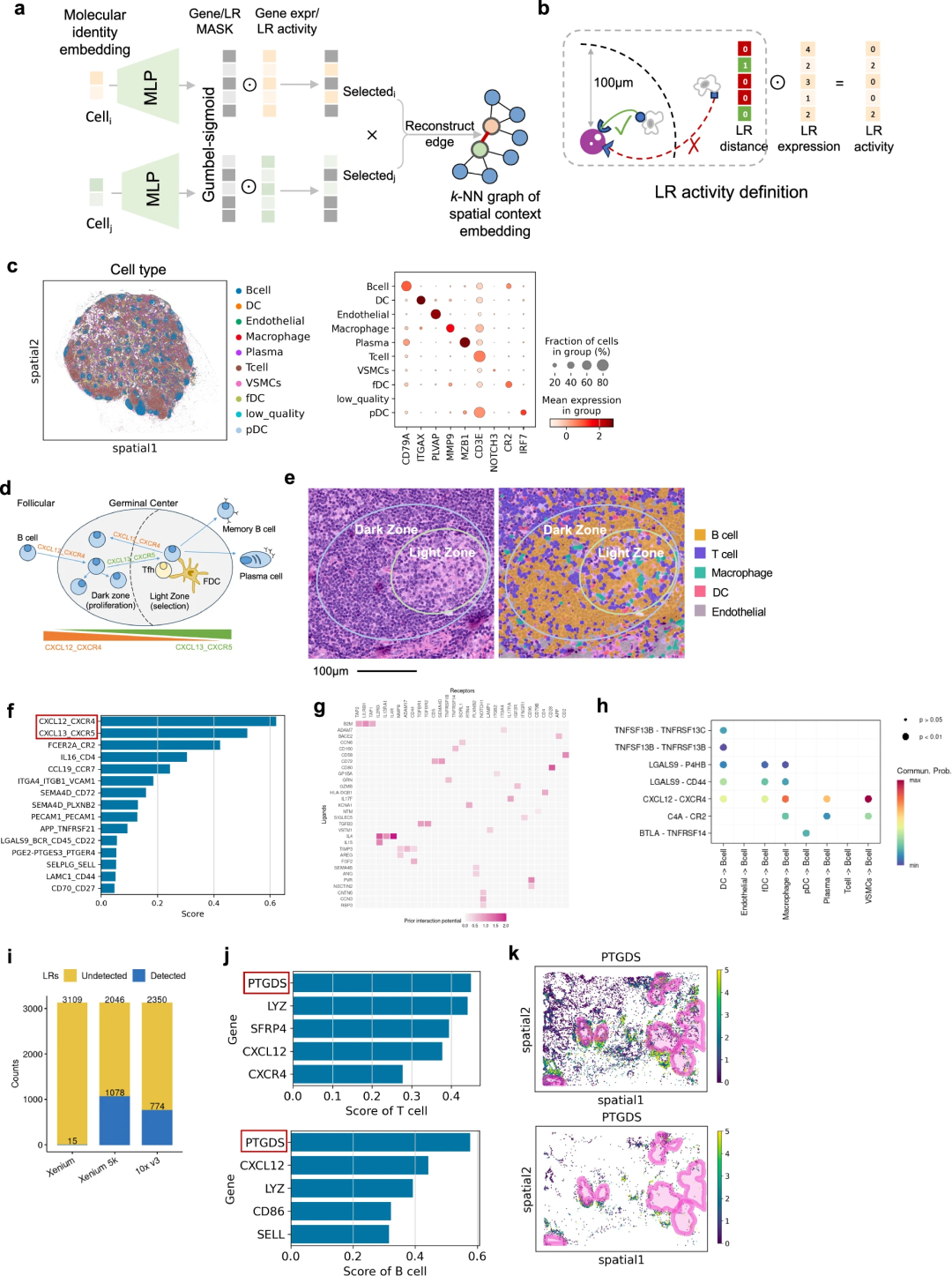
图3. DECIPHER能够识别与定位相关的LR对/基因
技术进步使得生成超过数百万个细胞的异质性空间组学图谱成为可能,这对计算方法的可扩展性提出了重大挑战。接下来,研究团队展示了DECIPHER在超大规模空间组学数据解析中的突破性能力。
在包含870万个细胞、涵盖8种不同癌症类型的人类泛癌空间图谱中,DECIPHER不仅成功校正了切片间存在的显著批次效应、实现了细胞类型的精准区分;还捕捉到癌症组织中细胞类型密度的连续变化,识别出肿瘤核心、淋巴低浸润肿瘤区、淋巴高浸润肿瘤区等三类肿瘤生态位。进一步分析发现,CCR7、CCL5与T细胞定位显著相关,并分别与初始T细胞激活和T细胞耗竭有关。值得注意的是,DECIPHER还具有极高的效率,单GPU运行时间不足4小时。
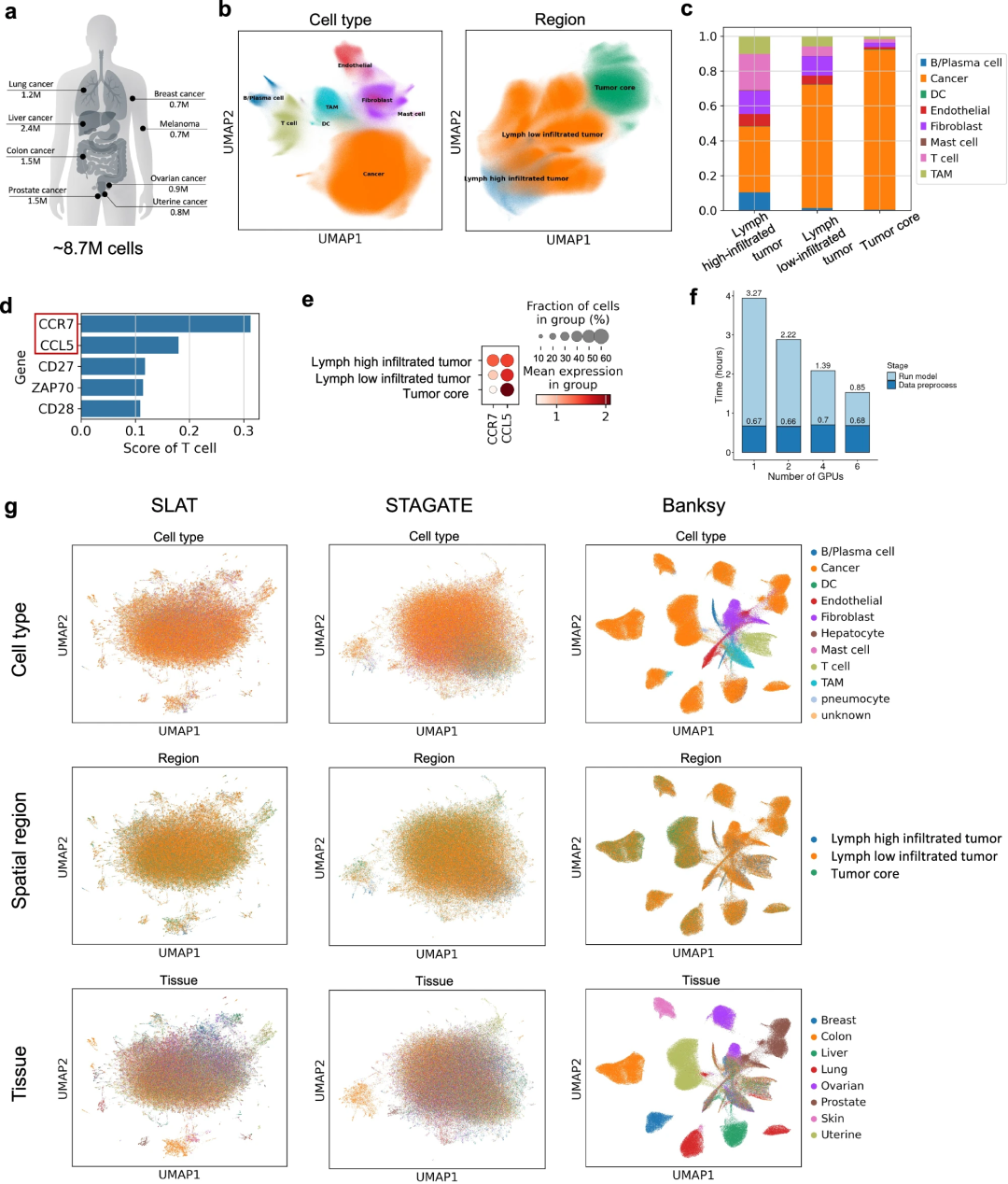
图4.人类泛癌空间图谱解析
DECIPHER还支持3D空间数据分析。在包含151个连续切片、共350万个细胞的小鼠全脑3D图谱中,DECIPHER生成的空间位置嵌入能精准对应艾伦脑图谱解剖区域,准确还原不同脑区细胞的空间分布,这凸显了3D信息对揭示复杂组织空间模式的必要性,以及DECIPHER在该场景下的优势。总之,该模型在大规模、高复杂度空间数据的处理与建模中,展现出远超主流方法的可扩展性、准确性与实用性。
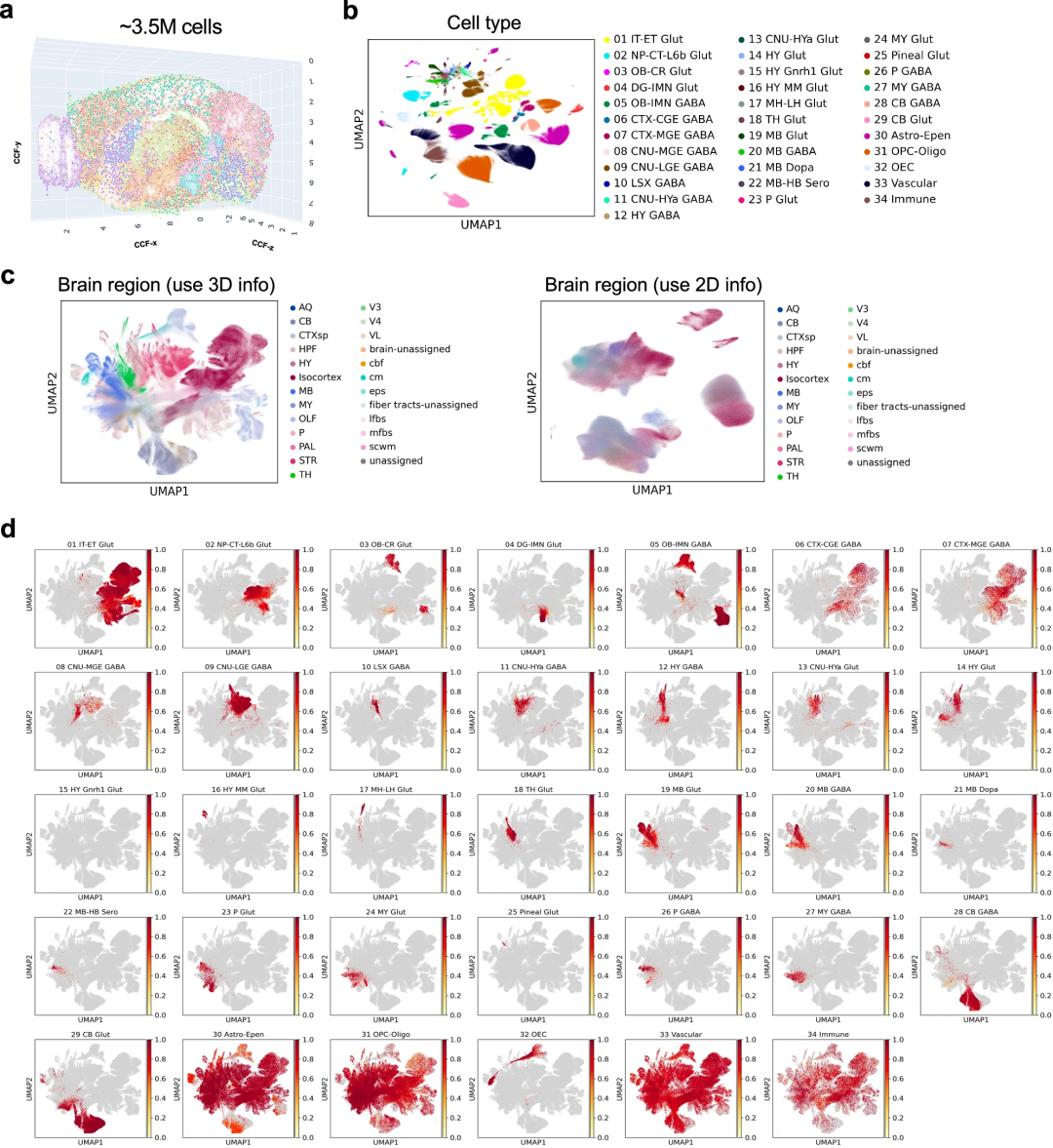
图5. 小鼠3D脑图谱分析
综上所述,DECIPHER为解决空间组学数据中细胞内与细胞外因素纠缠的难题提供了创新性的计算框架;其解耦嵌入不仅提升了模型性能与可扩展性,还增强了对细胞–微环境互作机制的解析能力。该模型助力人们更深入探索细胞功能及其在生理、病理状态下的调控机制,有望推动发育、免疫及肿瘤等领域在空间背景下的精细研究。
DECIPHER代码:
https://github.com/gao-lab/DECIPHER
参考文献:
Xia, CR., Cao, ZJ. & Gao, G. DECIPHER for learning disentangled cellular embeddings in large-scale heterogeneous spatial omics data. Nat Commun 16, 7991 (2025). https://doi.org/10.1038/s41467-025-63140-8


